
大数据文摘出品
作者:千雪、曹培信
如果你被隔离在家,陪伴你的只有一台电脑,你会做什么?
打游戏、看剧或是在线吃瓜?
也许帮助抗击新冠疫情会更有意义一些,通过一个叫做Folding@Home项目,你可以利用自己个人电脑的空闲运算资源帮助预测SARS-CoV-2蛋白质的3D形状,从而为寻找可以治疗这种疾病的药物出一份力。

Folding@Home:
https://foldingathome.org/
文摘菌也去助力了一把,下载软件安装之后,就是一个简洁的页面,可以选择你支持的研究,文摘菌选择的是COVID-19,也可以选择助力的程度,选择Medium基本不影响电脑使用,但是选择Full会让电脑有点卡。
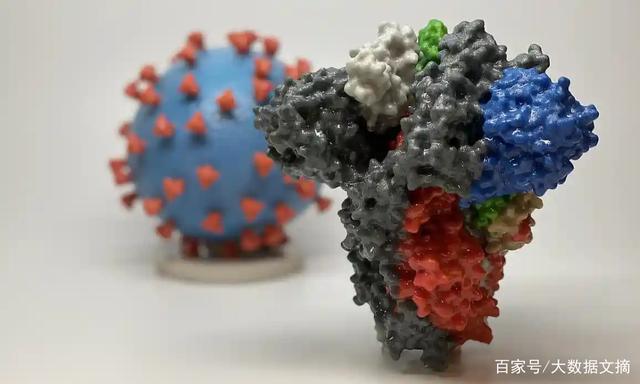
参与者可以在家用电脑上帮助“折叠蛋白”
早在1977年,生物学家Jean和Peter Medawar就提出病毒“只是包裹在蛋白质中的一条遗传信息”,病毒必须感染进入活细胞才能进行复制和传播。
Folding@Home的研究人员Greg Bowman表示,SARS-CoV-2病毒的蛋白质主要用来抑制我们的免疫系统和自我繁殖,为了帮助对付SARS-CoV-2病毒,我们需要了解这些病毒蛋白是如何工作的,以及我们如何设计治疗方法。
那普通人如何能帮助了解蛋白质呢?
首先我们要知道,蛋白质是由一系列被称为氨基酸的化学物质组成的,在多数情况下,这些化学物质会自发地“折叠”成紧密的功能性结构,就像一台机器一样,决定蛋白质功能的是蛋白质成分的排列和移动方式。
确定蛋白质结构的实验方法很多,但它们一般只能揭示蛋白质通常形状的一个特定时刻的形状,但是蛋白质一般是运动的状态,在实验方法中错失的结构可能是发现新疗法的关键。
就像橄榄球比赛一样,一般的实验只能看到刚开场是球员摆列整齐的状态,而我们需要了解的是整个比赛中所有人的运动状态。

这时候计算机模拟就上场了,我们可以通过计算机模拟来理解蛋白质的运动。计算机模拟蛋白质结构的过程就被称为“折叠蛋白”,但是这个计算量是巨大的,蛋白质可以在每个氨基酸之间扭曲和弯曲,所以一个含有数百个氨基酸的蛋白质有可能具有数量惊人的不同结构:数量级是1后面跟着300个零。
超级计算机也很难进行这样的运算,于是Folding@Home就发起了针对SARS-CoV-2蛋白质的分布式计算项目,普通人在家中可以将自己个人计算机的计算资源贡献出来,帮助进行“折叠蛋白”的模拟。
目前已有一百万人参加,运算能力=500台超级计算机
该项目如今已得到超过100万人的响应,根据运作该分布式计算工作的团队Folding@Home的说法,3月25日,该计算机的网络联合计算能力就达到了每秒操作1,000,000,000,000,000,000次,即1百亿亿次。
这使其功能比目前世界上最快的传统超级计算机IBM Summit还强大了六倍,到了上周一,该项目的计算能力又翻了一倍还多,刷新了2.4百亿亿次的新记录,比前500台传统超级计算机的总和还快。
这反映了全球的志愿者们对Folding@Home项目的巨大支持,大家只要用电脑下载运行一个简单的软件,就可以在家完成一些小任务来帮助确定蛋白质的3D物理结构。
软件下载地址:
https://download.foldingathome.org/releases/public/release/fah-installer/windows-10-32bit/v7.6/fah-installer_7.6.9_x86.exe
了解蛋白质结构,将有助于开发抑制药物
通过对蛋白质运动结构的模拟,可以寻找蛋白潜在的“药物结合点”,从而开发出可以抑制病毒蛋白质的药物。
在Folding@Home的最新研究中,他们模拟了一种来自埃博拉病毒的蛋白质,这种蛋白质通常被认为是“不可受药”的,因为实验中的蛋白质状态没有明显的可药物位点。
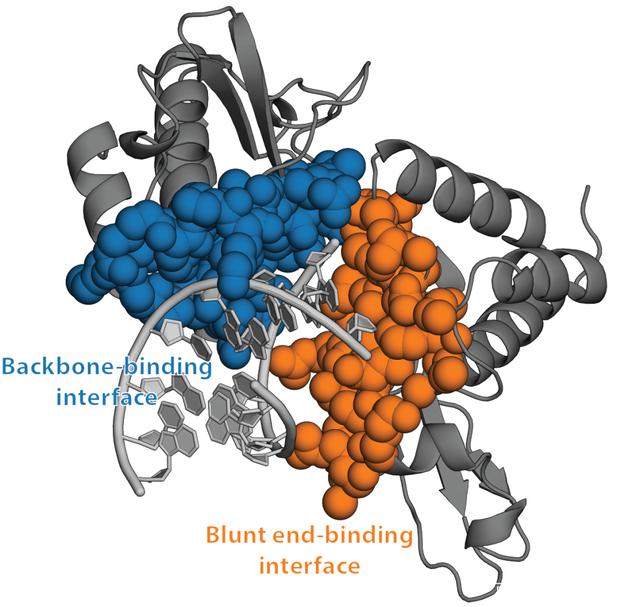
埃博拉病毒蛋白质的实验结构没有明显的药物作用位点(球状的原子间没有深的“口袋”)
但是,Folding@Home通过计算机模拟发现了另一种结构,确实有一个可药物的点。重要的是,研究人员随后进行了实验,证实了我们的计算预测,目前他们正在寻找与这个新发现的结合位点相结合的药物。
现在对于SARS-CoV-2研究的目的也是如此。
现在,这项研究已经得到了初步回报。Greg Bowman说,他们的其中一项工作就是持续关注SARS-CoV-2用于入侵人类细胞的“刺突蛋白”,刺突必须经过剧烈的张口运动,才能揭开细胞表层,最终与人类细胞进行结合,了解刺突究竟是如何打开的,这会非常有用,毕竟治疗过程中的每一个步骤都可能是针对性治疗。
“然而,至少以现有的实验技术,我们还无法观察到刺突是如何发生这种变化的,刺突张口状态的数据也很有限。”但是仅仅几个星期之后,这个团队就通过该项目创建出一个仿真模型,模拟了刺突张开“嘴巴”的第一阶段。
被比特币带歪的“全球共享分布式计算”项目
用强大的计算力对蛋白质的结构进行模拟显然是一个非常有意义的项目,而号召全球的志愿者共享出自己的个人计算机,更是彰显了人类在抗击疫情中的团结合作。
但是这样的项目的生存空间却因为“挖矿”而越来越小。
比特币和加密货币的兴起抑制了Folding@Home和类似项目的发展,毕竟“挖矿”为空闲的个人计算资源提供了另一种不那么无私的用途:“开采比特币”就可以挣到真正的钱。
就在Folding@Home发布Covid-19相关新任务的前不久,分布式计算工作的先驱项目SETI@home被关闭。
自1999年以来,SETI@home一直向世界各地的计算机发送射电望远镜数据,然后对这些数据进行分析,从而寻找外星生命的迹象。但是上个月,研究人员关闭了该计划。
SETI@home的组织者说:“管理数据的分布式处理对我们来说是个很麻烦的工作。我们现在需要集中精力对已有结果进行后端分析,然后发布在科学期刊中。”
相关报道:
https://www.theguardian.com/technology/2020/apr/15/volunteers-create-worlds-fastest-supercomputer-to-combat-coronavirus
https://www.theguardian.com/science/2018/dec/02/google-deepminds-ai-program-alphafold-predicts-3d-shapes-of-proteins
https://foldingathome.org/about/the-foldinghome-consortium/
- 本文固定链接: http://www.qiantao.net.cn/?id=721
- 转载请注明: admin 于 千淘万漉 发表
《本文》有 0 条评论